轉座子(TEs)是真核生物基因組中廣泛存在的DNA重復序列,約占水稻基因組的35%。轉座子是植物產生遺傳變異的重要來源,通過多種機制調控基因表達及表型變異。水稻的泛轉座子變異圖譜研究表明,轉座子在水稻馴化和育種性狀改良方面發揮重要作用。
近日,中國科學院院士、遺傳與發育生物學研究所研究員李家洋帶領的科研團隊,聯合崖州灣國家實驗室的研究人員,在《植物生物技術雜志》(Plant Biotechnology Journal)上在線發表了題為Generation of OsGRF4 and OsSNAC1 alleles for improving rice agronomic traits by CRISPR/Cas9-mediated manipulation of transposable elements的研究論文。該研究通過對水稻基因OsGRF4或OsSNAC1的非編碼區進行轉座子編輯,實現了對目的基因表達的精確調控。同時,該研究創制的優良等位基因為作物遺傳育種提供了新策略。
微型反向重復轉座子(MITEs)是短小而非自主的DNA轉座子,是水稻基因組中數量較多的轉座元件,且與至少58%的水稻基因相關。研究表明,MITEs是水稻基因表達變異的主要驅動因素之一,而利用MITE插入多態性進行全基因組關聯研究有助于挖掘并控制農藝性狀的潛在基因。
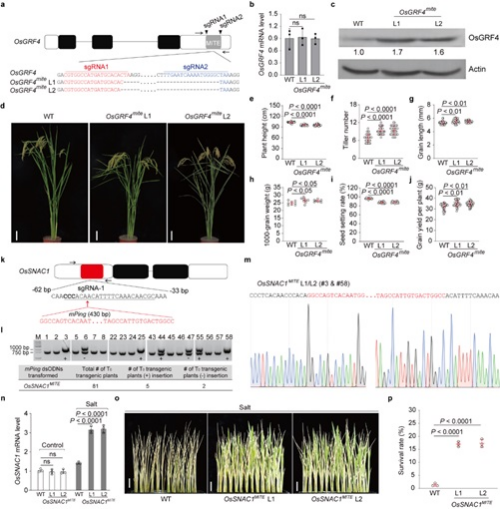
該研究推測,通過CRISPR/Cas9基因編輯技術設計基因非編碼區的MITEs轉座子分布可以上調或下調目標基因的表達,從而創制新的基因等位基因型以改良水稻性狀。為驗證這一設想,科研人員選擇水稻中的生長調節因子4基因OsGRF4和脅迫響應基因OsSNAC1進行研究。研究顯示,OsGRF4可正向調控水稻產量的相關性狀,在其終止密碼子下游的1200bp內插入一個294-bp的PIF/Harbinger超家族MITE;OsSNAC1可以增強水稻的耐鹽性,但在其上下游非翻譯區未檢測到MITE。研究發現,水稻某些基因下游非編碼區中的MITE可以介導靶基因的翻譯抑制。因此,研究認為,通過CRISPR/Cas9技術刪除OsGRF4下游非翻譯區中的MITE,可以創制出過表達的等位基因型。研究針對OsGRF4基因的MITE靶區域,設計構建了2個CRISPR/Cas9 sgRNAs,并對其進行編輯。科研人員對得到的無轉基因的純合突變體進行分析發現,OsGRF4基因的MITE刪除,提高了OsGRF4mite突變體中靶蛋白的豐度,并改善了與產量相關的農藝現狀。水稻基因上游非翻譯區中的一些MITEs可作為增強子,如miniature Ping (mPing) TE可以增強鹽脅迫響應基因的轉錄水平。因此,研究人員嘗試將430-bp的mPing插入耐鹽基因OsSNAC1的上游非翻譯區。進而,科研人員剖析得到的純合突變體發現,OsSNAC1基因的MITE插入,提升了鹽脅迫下OsSNAC1MITE突變體中靶基因的轉錄水平,并增強了它的耐鹽性。
上述成果為轉座子驅動的作物遺傳育種提供了新途徑。
研究工作得到科技創新2030-重大項目、國家自然科學基金及海南省相關項目的支持。
通過基因編輯技術設計非編碼區的MITEs轉座子分布來改良水稻性狀